168 views
owned this note
[TOC]
# Science Ouverte pour les Sciences Humaines et Sociales : scripts, codes et logiciels
*22-27 juin à Saint-Pierre d'Oléron*
## Introduction
### Ressources utiles
| Nom | URL |
| ----------- | ----------------- |
| Revue Rzine | https://rzine.fr/ |
| MOOC | https://www.fun-mooc.fr/fr/cours/recherche-reproductible-principes-methodologiques-pour-une-science-transparente/ |
| GT Notebook| https://gt-notebook.gitpages.huma-num.fr/site_quarto/ |
- **MOOC "Recherche reproductible"** (Arnaud Legrand) :
- Non "technique", en Français
- Module 1 = prise de notes, format texte, recherche d'information, utilisation basique de gitlab
- Module 2 = document computationnel (aka notebook) avec 3 technologies
- Jupyter
- Rstudio
- Org-mode
- Module 3 = rédiger une analyse reproductible (du téléchargement des données aux figures)
- Module 4 = les enfers de la recherche reproductible (i.e., les limites de ce que l'on vient de présenter)
- **MOOC Reproductible Research II :** https://www.fun-mooc.fr/fr/cours/reproducible-research-ii-practices-and-tools-for-managing-comput/
- Module 1 : gestion de données
- Méta-données et formats de données "avancés" par rapport à csv: JSON, FITS, HDF5
- Git-Annex: la **bonne** extension à git pour gérer des données scientifiques
- Archivage (Zenodo, SWH)
- Module 2 : gestion d'environnements logiciels
- En quoi ça consiste compiler/installer un logiciel
- Utiliser Docker pour exécuter du code de façon controlée sans casser sa machine
- Identifier ses dépendances et construire un environnement logiciel de façon reproductible
- Docker/Singularity/Guix
- Module 3 : gestion de calculs complexes
- Notion de workflow en comparaison à la vision script ou notebook
- Illustration avec Make et Snakemake (Note for R users: https://workflowr.github.io/workflowr/ does not seem to correspond to the same notion)
- **GT Notebook :** https://gt-notebook.gitpages.huma-num.fr/site_quarto/
## Les projets en groupe
### Constitution des groupes
- Des groupes de 4-5 personnes: la diversité, c'est la vie! :stuck_out_tongue_winking_eye:
- 7 groupes de 4, 1 groupe de 5
- Au moins une personne avec un linux ou un macOs dans chaque groupe
- Un seul langage par groupe
- Au moins une personne qui est à l'aise avec git
- Définir "être à l'aise" ? = git clone, git add, git commit, git checkout, git pull, git push, git __merge__ ? :+1:
Si vous avez déjà "mergé" des branches, vous êtes à l'aise avec git.
### Le dépôt du projet
Le repo du projet est ici : https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/olympix
## Open science
### Lundi 11h - Laurence El-Khouri - Direction des Données Ouvertes de la Recherche (DDOR)
Présentation et organigramme [Lien](https://www.science-ouverte.cnrs.fr/ddor-cnrs-direction-des-donnees-ouvertes-de-la-recherche/)
- La DDOR et le paysage général:
- pas mal de gens passés par les instances de direction du CNRS sont maintenant au Ministère
- Proche de la DGDS et des 10 instituts
- Directrice, COPIL, DAS de chaque institut dédiés à Données Ouvertes et Science Ouverte
- 3 unités rattachées: CCSD (HAL et autre), INIST (Plans de Gestion de Données, identifiants permanents), PERSEE (portail de mise à disposition revues non numériques et TAL Traitement automatique des langues)
- Depuis 2017: Marin Dacos (ancien directeur d'OpenÉdition) a pris en charge la SO au Ministère :
- 2018: PNSO, Ouvrir la science
- 2021: PNSO 2, politique des algorithmes et des codes sources
- Vont devoir redoubler d'attention avec ce qu'il se passe aux US.
- Actuellement à la manœuvre: Isabelle Blanc (AMDAC, Administrateurs Ministériels des Données, des Algorithmes et des Codes sources)
- [Feuille de route science ouverte CNRS (2019)](https://www.cnrs.fr/sites/default/files/press_info/2019-11/Plaquette_ScienceOuverte.pdf)
- Objectif = 100% d'accès ouvert, ouvert autant que possible, fermé autant que nécessaire
- DDOR fait des synthèses (où on en est, par rapport au reste des universités et à l'Europe), des recommandations, et essaie de construire un consensus au moins au niveau des concepts
- Dépôt d'articles des chercheur·euse·s dans HAL institutionnalisé, donc on est à 95% de dépôt en accès ouvert au CNRS
- Que s'agit-il d'ouvrir ?
- Article 30 pour la Loi pour une République numérique (6 à 12 mois d'embargo sur le MAA)
- Quel que soit le contrat que vous avez signé avec quiconque, si vous êtes payé par de l'argent public, vous avez le droit de déposer la version auteur dans une archive ouverte, et ce même si vous avez cédé votre copyright
- Reco 1 : ne jamais céder son copyright (droit inaliéable, *même si en pratique, c'est compliqué*) mais plutôt le partager avec l'éditeur
- Reco 2 : regarder les demandes de l'éditeur et les licences qui seront aposées
- Les 50 nuances d'accès ouvert: immédiat/différé, payant pour celles/ceux qui mettent à disposition, etc.
- 
- « Find money, be famous or vanish »
- 
- [Modèles Diamant](https://www.unesco.org/en/diamond-open-access) ou dépôt en archive ouverte : Certains éditeurs commerciaux (Nature) demandent jusqu'à 9000€ d'APC pour publier les articles en accès ouvert et "en moyenne", on est à 3000€. *Le prestige, c'est quelque chose qu'on construit nous-mêmes et qu'on accepte...*.
- Les APC (droits à publier), par an, représentent environ 4 millions € pour le CNRS;
- BibCNRS (outil du CNRS qui permet l'accès à des articles non ouverts): ça représente paie 13M€ par an pour les abonnements et 4M€ pour les APC. À l'échelle française, on est à 100M€.
cf. [études Paolo Crosetto](http://openscience.ens.fr/ABOUT_OPEN_ACCESS/ARTICLES/2024_11_08_Quantitative_Science_Studies_The_strain_on-scientific_publishing.pdf)
- État des lieux de la publication scientifque: pesque doublement des publications scientifiques en 10 ans, avec raccourcissement des temps de relecture, revues prédatrices (MDPI, Frontiers, ...) qui innondent le marché de numéros/journaux spéciaux, des papermills qui génèrent des articles, etc.
- Impact Factor ~ [Effet cobra](https://fr.wikipedia.org/wiki/Effet_cobra)
- Loi de Goodhart : « Lorsqu’une mesure devient un objectif, elle cesse d’être une bonne mesure. »
- Loi de Campbell : « Plus un indicateur social quantitatif est utilisé comme aide à la décision en matière de politique sociale, plus cet indicateur est susceptible d'être manipulé et d'agir comme facteur de distorsion, faussant ainsi les processus sociaux qu'il est censé surveiller. »^[Campbell, « Assessing the impact of planned social change », Evaluation and Program Planning, vol. 2, no 1, 1979, p. 67–90]
- [Retraction watch](https://retractionwatch.com/) : site qui recense les articles rétractés d'une revue. [Problematic paper screener](https://dbrech.irit.fr/pls/apex/f?p=9999:1::::::) = logiciel développé par Guillaume Cabanac.
- Discussion sur les services utilisés par tous (arxiv, etc.) et leur soutien par les institutions. Au CNRS, les 500K€ servent à soutenir Arxiv, Mersenne, PCI, SciPost.
- Quand vous utilisez quelque chose de gratuit, assurez-vous d'y contribuer en retour. Même principe pour les articles (Si vous publiez 1 article, reviewez-en au moins 2) ou les données (participez à la curation de données).
- Ancré dans les problématiques d'évaluation de la recherche, PlanS, [COARA](https://coara.eu/), etc. pour s'éloigner des pratiques quantitatives de bibliométrie et apprécier qualitativement l'activité des chercheur·euses dans toute leur diversité.
- Open Science = An Antidote to Anti-Science (surtout dans le contexte politique internationnel actuel)
- Méthode des [3Rs](https://nc3rs.org.uk/who-we-are/3rs) : Replacement, Reduction and Refinement
- Actuellement gros efforts sur la publication systématique des travaux expérimentaux et des données en lien avec l'expérimentation animale.
- DDOR: 3 unités rattachées
- **INIST:** évoque l'extention [Click & Read](https://clickandread.inist.fr/) qui indique, quand on est sur un article Wiley ou autre, les alternatives en accès libre
- **OPIDOR:** infrastructures numériques recherche et service (DMP Opidor, [Cat OPIDOR](https://cat.opidor.fr/index.php/Cat_OPIDoR,_wiki_des_services_d%C3%A9di%C3%A9s_aux_donn%C3%A9es_de_la_recherche), PID OPIDOR)
Et des Actions Nationales de Formation à la données, science (par ex: https://www.science-ouverte.cnrs.fr/actualite/anf-tdm-2024-inscriptions-ouvertes/)
CNRS research Data dans https://entrepot.recherche.data.gouv.fr/dataverse/cnrs
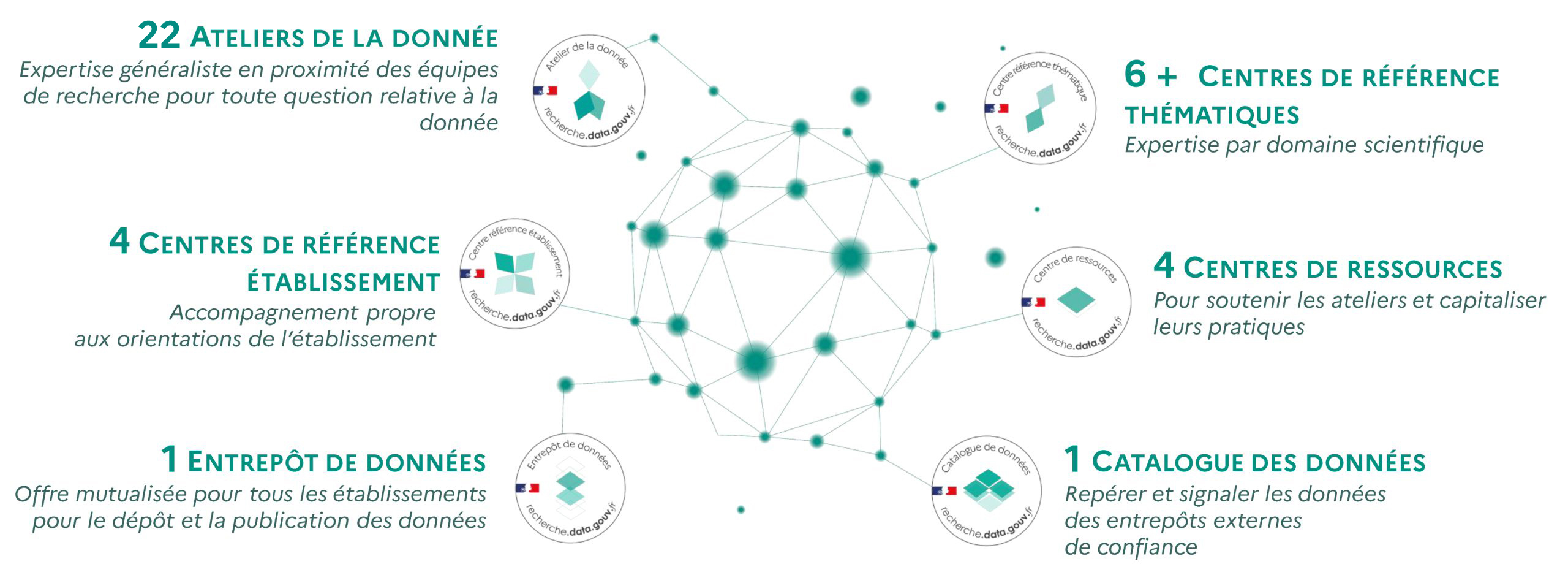
- Les **Ateliers de la données :** 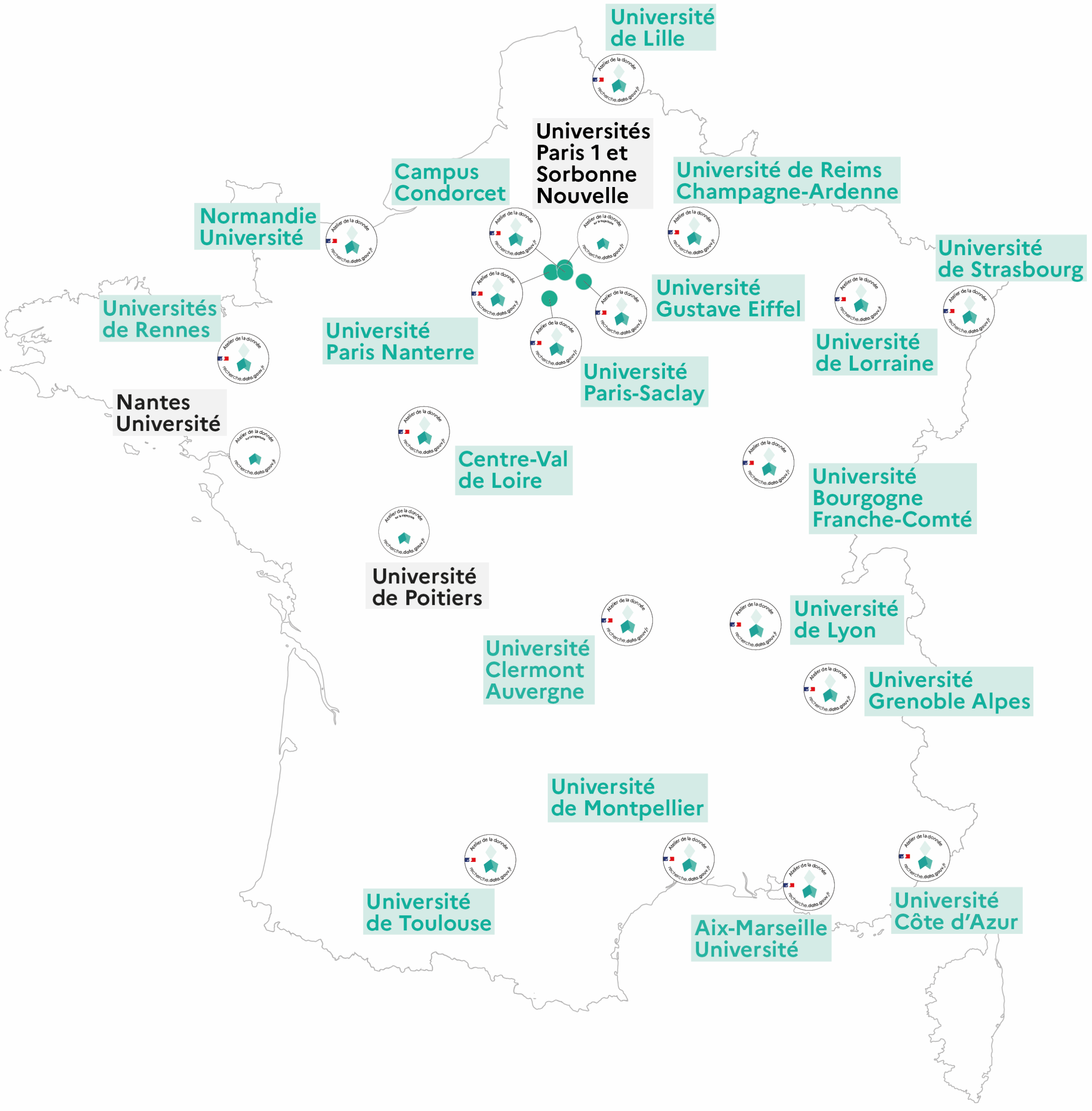 *NB : La création des Ateliers de la donnée est à l'initiative des établissements, notamment des Universités - il faut répondre à un appel. Calendrier des prochains appels : [lien](https://recherche.data.gouv.fr/fr/page/appels-a-manifestation-dinteret-ateliers-de-la-donnee)*
- **Research Data Alliance:** hub mondial permettant d'avoir accès à une grande diversité et d'identifier les groupes de travail, communautés de pratiques, etc. susceptibles de nous intéresser: http://www.rd-alliance.org/group-directory
- Reproducibility Checklist Working Group
- Alignement of multilingual vocabulary in SHS
- Grosse préoccupation en ce moment sur les données sensibles/données de santé
- Health Data Hub
- Habilitation à l'hébergement de données en santé
- Le CNRS bénéficie d'un accès permanent au SNDS (Système national des données de santé)
**Questions**
- <span style="color:green">Beaucoup de choses sur les données de la recherche, mais que fait-on pour les modèles d'IA ? On est entre code et données ? (et Git n'est pas satisfaisant pour des modèles de grande taille)</span>
- *R : Pas d'espace de stockage dédié aux LLM. Des choses ([Bloom](http://www.idris.fr/annonces/hawards-hpc-wire-2022-eng.html)) côté IDRIS (Institut du développement et des ressources en informatique scientifique). Il y a une newsletter de l'IDRIS: [panoram'IA](http://www.idris.fr/panoramia.html) qui est très bien. Voir aussi [ComparIA](https://www.comparia.beta.gouv.fr/) du ministère qui vous permet de prendre conscience de l'impact de ces services (notamment environnemental) tout en évaluant la pertinence de leurs réponses.*
- *C'est un des défis identifiés par le CNRS. [PRAIRIE](https://u-paris.fr/projet-prairie-institut-interdisciplinaire-dintelligence-artificielle-3ia/) travaille aussi là-dessus.*
- <span style="color:green">Les économistes sont mauvais en SO mais très bons avec ChatGPT et utilisent ça pour générer des rapports, ce qui pose des problèmes de souveraineté. Des alternatives chez huma-num ?</span>
- *R : Au CNRS, il est interdit de payer un abonnement ChatGPT sur des budgets CNRS (mais il peut être payé par d'autres budgets...). Dans un cadre de changement culturel, c'est compliqué de convaincre.*
- *R : Alternatives à proposer: on est en retard, on développe des solutions internes mais elles ne sont pas encore aussi bonnes car on n'a pas les mêmes moyens. Mistral.ia n'est pas totalement ouvert (c'est français mais quand même financé par de grands groupes internationaux). Même problématique pour l'abandon de l'impact factor (en particulier avec la section d'économie). Il faut tenir compte des temporalités différentes d'une communauté à l'autre mais il faut prendre son mal en patience et garder espoir.*
- <span style="color:green">On organise un Workshop promouvant les nouvelles bonnes pratiques en lien avec l'IA. Comment publier ? En économie, on publie les travaux quand ils sont finis, pas les travaux en cours. D'autre part, la publication dans des revues ouvertes n'a aucun impact sur les carrières donc on manque d'arguments pour activer la transformation.</span>
- *R :*
- *Commentaire : en formation d'intégrité de la recherche [DOCTI-SCOPE](https://isblue.fr/actualites/doctis-cope-formation-doctorale-integrite-scientifique-comment-publier-de-maniere-integre/) (Formation doctorale & intégrité scientifique : comment publier de manière intègre ?) il n'est pas recommandé aux doctorants de s'orienter vers les revues diamant, et de laisser la place aux chercheur.e.s dont ça n'impacterait pas la carrière.*
- <span style="color:green">La science ouverte, c'est bien , mais comment garantit-on la qualité. XVIII-XIXè = science faite par une minorité privilégiée qui "garantit" la qualité. Au XXè, démocratisation, et sortie de la dynamique de prestige pour une science très large et en lien avec les citoyens, ce qui ouvre des tensions.</span>
- *R : La qualité de la donnée n'est pas non plus forcément garantie dans Nature. Beaucoup d'articles sont retractés dans les revues dites de bonne qualité. Aujourd'hui, on compte sur le peer review, mais il n'est pas infaillible. Pas de vérité absolue en science, qui va forcément évoluer. Il faut se fier au fait que la donnée a été reviewée*
- *Commentaire : communauté [Peer Community In](https://peercommunityin.org/) et la revue associée. Voir aussi : https://peercommunityjournal.org/. Autre platinum open-access peer-reviewed journal [ReScienceC](https://rescience.github.io/) qui cible la recherche computationnelle.*
- <span style="color:green">Spécificité Données pour les SHS (MSH, ateliers de la donnée, ...)</span>
- *R : le CNRS/INSHS et la MSH participent aux ateliers de la données. Le CNRS est impliqué dans le GT5 (https://www.reseau-capteurs.cnrs.fr/gt/gt-documentation-des-donnees-issues-de-capteurs/) (logiciel), évoque Violaine Louvet, Christine Hadrossek, Olivier Rouchon, ...*
- <span style="color:green">Sur les aspects de sobriété énergétique, on incite les chercheurs à déposer leur données dans des entrepôts. Stockage exponentiel. Que stocke-t-on ? Ou pas ? Quelles sont les bonnes pratiques ?</span>
- *R : [RIP in data](http://corist-shs.cnrs.fr/node/1600), c'est encore une question ouverte qui dépend beaucoup du contexte.*
- <span style="color:green">Des projets pour remplacer ces revues "prestigieuses" ?</span>
- *R : Oui, il arrive que des comités de chercheurs quittent les comités éditoriaux de journaux d'Elsevier, Springer, etc. mais le panorama de ce mouvement n'est pas encore établi. C'est courageux, ça implique de tout reconstruire car l'éditeur reste souvent propriétaire du titre mais on observe des transferts d'une revue à son homologue ouvert.*
- *Infrastructure qui aide à publier, recommandée par le CNRS : https://scipost.org/*
- <span style="color:green">Est-ce que le CNRS va prendre en compte l'implication dans le mouvement "science ouverte" (publications dans les revues ouvertes...) pour les recrutements ?</span>
- *R d'Arnaud Legrand : d'après mon expérience en section 6 : une orientation est donnée par le CNRS, mais c'est à chaque session de s'organiser. Ce qui va compter, ce sont les recommandations que chaque session va mettre sur son site*
- *R : orientations au CNRS : 1) s'intéresser davantage à la qualité qu'à la quantité des publications ; 2) élargir le type de résultats sur lequel le chercheur peut s'appuyer. D'ailleurs, l'attribution des "médailles" du CNRS ne se base pas (plus ?) sur la quantité des publications*
### Lundi 14h - Kamran Naim (CERN) : Accelerating Open Science
Présentation de l'Open Science au sein du CERN : [lien](https://openscience.cern/)
*Pour l'anecdote : ils produisent plus de données qu'il n'existe de capacité de stockage*
Motivation de l'open science en physique : rendre la discipline accessible à tous.tes, parce que champ de la recherche complexe
Politique open data au CERN n'est pas imposée. Ils la rendent possible (?)
**Open Access**
- Accord avec les principaux journaux scientifiques de la discipline : on s'abonne à vos publications, et on a des accords sur la publications open access
- Différents mécanismes : [SCOAP3](https://openscience.cern/node/433), modèles collectifs, OA agreements, individual APC
- [SCOAP3](https://scoap3.org/) : partenariat fondé en 2014, regroupant plus de 3000 institutions de recherches, bibliothèques, organismes de financements, dans 44 pays, régions ou territoires. En vue d'ouvrir la publication de la recherche
Pourquoi payer pour faire de la recherche ? Pourquoi payer pour lire la recherche. Problème moral
- [Déclaration de Budapest](https://www.budapestopenaccessinitiative.org/read/): le savoir scientifique doit être un bien commun
- Liberate scientific research and make it available to the whole world, make it a public good and not a commodity. Unfortunately, it (articles) remained treated as a commodity and the barrier was enshifted to authors and did not turn into a public good
- Objectif SCOAP3 = really make it a public good by working with partner countries.
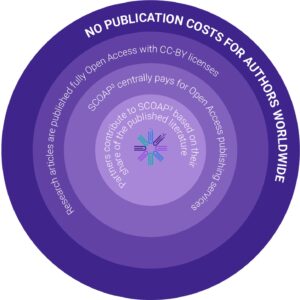^[SCOAP3 Consortium, https://scoap3.org/what-is-scoap3/]
- Look a 2-year period, look at all the research published in our discipline in the main journals (about 15000 papers), look at every single paper and authors and identify their country and affiliation. Si bi-affiliation sur plusieurs pays, assignment au pays avec le plus grand PIB.
- Permet d'évaluer quelle est la contribution effective de chaque pays (e.g., China moved from 8% to 12%) qui contribue monétairement en proportion via SCOAP3 (aux abonnements, du coup ???). Chaque pays, région ou territoire contribue à la mesure de sa production scientifique dans ce domaine.^[https://scoap3.org/what-is-scoap3/]
- 1200€ on average per article to make it available to the world, which is much lower than the average 4000$ of e.g., Physical Review Letters.
- Provide stable prices for all partners, regardless of thei local situation.
- This is achieved through a real leveraging power obtained by our consortium.
- Phase 4 SCOAP3 : de l'Open Access à l'Open Science
- Identification des éléments techniques de l'Open Science que les éditeurs peuvent adopter. Définition des termes et mise en commun au sein d'un glossaire. Ex:
- accessibility : notamment pour les personnes en situation de handicap
- data linked
- peer review
- standard methods
- Pénalités financières pour les éditeurs qui ne respectent pas ces méthodes/standards
- Méthode d'évaluation des éditeurs : [lien](https://scoap3.org/journals-2025-2027/open-science-elements/)
**Open data**
4 levels of data
- 1st = Data related to papers : Collaboration with the University of Durham. Open all the data related to a final publication
- 2 : Curated, containerized datasets
- 3 : données reconstruites et simulées pour les analyses scientifiques
- 4 : format brut, les données qui ne peuvent pas être stockées en l'état.
Open data policy published in 2020 and relates mainly to level 3 data. Data policy is a little bit to conservative, but we can look at it like a start. [see more](https://opendata.cern.ch/docs/cern-open-data-policy-for-lhc-experiments)
Researchers want to have the right to use that data first. Not an embargo, but some researchers are afraid being scooped (by collaborators...). Expectation that releases of datas should be delayed (5 years max)
If you want people to be able to extract data, it should be accompanied by
- reproductible samples
- reproducible operating environment (contextual resources)
Téléchargement de 1Terrabytes depuis la [plateforme d'opendata](https://opendata.cern.ch/) = impossible en local : solution ? Cloud ?
**Open softwares**
Invenio = open source software framework.
Powers Zenodo
Zenodo = world's largest research repository
INVENIO-RDM^[Research Data Management Platform] [introduction poster](https://zenodo.org/records/3484154)
**Open hardwares**
About the hardware development, there is a question : what is the most sociable (?) approach to take ? Do we open it ?
Reach the optimal social impact. Open technologies is not always the best way in that aspect.
**Incitations**
Le CERN a signé l'accord/agreement CoARA en 2022 + préparation du plan d'action de CoARA en 2024
Développement d'un cadre d'évaluation de la recherche
**CERN infrastructure**
Key infrastructure to enable open science. Range of different open infrastructure
- Open Data portal
- Analysis preservation
- [INSPIRE](https://inspirehep.net/) : open DataBase. Global index. Like an advanced Google scholar.
**Questions**
- "*Collaboration is the key*". How comes the CERN in the core of Europe promotes cooperation while competition is in the core of the foundation treaty of Europe ?
- *R: The competitivity values exist even at the CERN. People don't want to be scooped even by their own team. It is often about where we place our value. I think there are some aspects where there is an appropriate place to competition. But on key areas like science, when it comes to e.g. climate change, progress can only be reached with openness and collaboration.*
- Were the SCOAP<sup>3</sup> model game changer in the balance between research institution and the publishers ?
- *R: It has not been reproduced in other models. the cost of SCOAP<sup>3</sup> was quite high. Not every organization could have take an attempt. SCOAP<sup>3</sup> demonstrated that open access was possible, but not every organization has the possibility to apply it.*
- Collabodation is key, but so is money. What's in your opinion, the main obstacle to open science implementation.
- *R: Culture. We have this committee named the Scientific Infomation Policy Board and we had a conversation about Nature, with whom we had a "Gentleman agreement" saying that any paper published at CERN would be published open access. 5 years later, Nature changed this and charged us 9500€. There is a better way to spend this money but a paper in Nature is in Nature... We ended up joining the Swiss consortium, which had negotiated a limited number of paper fully open, but it turned out to be exhausted in April. Some researchers started gaming and submitting their paper in September, to that it would be in process by the end of the year and get a chance to benefit to the free charge before April. :face_palm:*
## Forges
### Mardi 9h - Franck Pérignon - Forges logicielles. Gestion de projets et développements collaboratifs. Gitlab, git et intégration continue.
> **Tout le monde doit demander un compte sur la forge GitLab du GRICAD.**
Pour s'inscrire sur la plateforme : https://register.gricad-gitlab.univ-grenoble-alpes.fr/
> Nous devions initialement utiliser le [GitLab de l'IR* Huma-num](https://gitlab.huma-num.fr/). Cette instance GitLab est celle qui nous est "naturellement" dédiée, c'est celle des SHS. Malheureusement, tout le monde n'a pu créer un compte avant l'ET. Nous utilisons donc celle du GRICAD.
> Les ressources de l'école sont donc ici: https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs
Le support est disponible ici en pdf : [https://so-shs.gricad-pages.univ-grenoble-alpes.fr/outils-collaboratifs-gitlab/cours/pdf/outils_collab_gitlab.pdf](https://so-shs.gricad-pages.univ-grenoble-alpes.fr/outils-collaboratifs-gitlab/cours/pdf/outils_collab_gitlab.pdf)
[Le site des cours](https://so-shs.gricad-pages.univ-grenoble-alpes.fr/outils-collaboratifs-gitlab/cours/)
[Des tps sont disponibles ici](https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/outils-collaboratifs-gitlab/trainings)
En complément du cours, pour la partie intégration continue décrite dans les slides, [des projets modèles, démos commentées sont disponibles ici](https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/outils-collaboratifs-gitlab/demos).
### Les forges
**Forges externes** = [Github](https://github.com) (racheté par Microsoft en 2019), alternatives [BitBucket](https://bitbucket.org) et [Gitlab.com](https://gitlab.com).
**Forges académiques** = Forge ESR (Enseignement Supérieur de la Recherche) comme https://gitlab.humanum.fr/, https://gricad-gitlab.univ-grenoble-alpes.fr/, ou https://gitlab.inria.fr/ qui sont toutes les trois des instances (déploiements) du logiciel GitLab (qui est open source) avec des comptes utilisateurs et des règles d'usage qui leurs sont propres.
### Git Premiers pas - commandes
```bash=
git config --global user.name "Prénom Nom"
git config --global user.email toto.tutu@my-univ.fr
git config --global alias.hist "log --pretty=format:'%h %ad | %s%d [%an]' --graph --date=short" # vous permettra de voir votre historique en faisant 'git hist'
git config --global alias.wdiff "diff --color-words" # vous permettra de faire des diff à la granularité du mot
# Gestion d'un projet en local
cd workdir && git init # Alternative git clone some_project_on_gitlab_or_github
git config --local user.name "Dark Vador" # Si vous voulez que, pour ce projet particulier, vos commits apparaissent dans l'historique comme signés par Dark Vador
git config --local user.email dark@vador.com
# do some stuff
git status # pour voir les fichiers trackés ou pas et leur état
git add somefile.txt some_other_file.md
git commit -m "J'ai rajouté mes deux premiers fichiers!"
# do some stuff
git status
git diff # pour voir le détail des différences
git diff --color-words
git commit --amend # rééditer le message du dernier commit réalisé
git add mon_fichier.txt
git commit -m "Des informations essentielles!"
# Ajout/Suppression de fichiers vides depuis ligne de commande bash
touch README.md # un fichier markdown vide est créé
rm nomdefichier.txt # suppression du fichier
rm 'nom de fichier.txt' ou "nom de fichier.txt" # les espaces dans les noms de fichiers sont lus
git status # on vérifie toutes les modifications apportées
git add . # si tout est ok : on ajoute le tout
git add nom_fichier.txt # OU on ajoute un fichier spécifique à commiter
git log # ou git hist si vous avez défini l'alias ci-dessus
git show 6f73c8913d849446474073e551b63f0f65fc7dbe # pour voir les modifications faites à chaque fichier dans ce commit particulier
```
> <span style="color:green">***Questions :** [name=Kim]En local, si j'ai deux répertoires avec deux identifiants de connection différents (Dossier A, ID A; Dossier B, ID B). Les identifiants de connection sont associés à chaque dossier ? Je n'ai pas besoin de me reconnecter avec `git config --global user.name` ? Si je navigue avec gitbash dans mes dossiers, il lira automatiquement les identifiants assignés au repo local ?*</span>
> > [name=Arnaud] Le `git config --global user.name` n'est **pas** un mécanisme de "connection" ou d'authentification. Ça indique juste au prograMme `git`, sur ta machine, que tout commit que tu vas faire à l'avenir sera annoté comme ayant été créé par le nom fournit. C'est une information uniquement utilisée pour la lisibilité de l'historique.
> > On pourrait imaginer que dans Dossier A, tu veuilles signer tes messages de commits avec "Jo" et dans le Dossier B, tu veuilles signer tes messages de commits avec "Jane". C'est possible, il faut juste faire un `git config --local user.name Jo` dans le dossier A et l'équivalent dans le dossier B.
> > <span style="color:green">*[name=Kim] Je vois ! Donc quand je naviguerai dans mes dossiers à l'avenir, avec gitbash par ex., je n'aurais plus besoin de reconfigurer ? A condition que `git config --local user.name` ait bien été spécifié dans chaque dossier.*</span>
> > [name=Arnaud] Oui, quand tu fais `config --global`, c'est valable pour **tous** tes dossiers (en fait, ça écrit l'information dans le fichier `.gitconfig` de ton home directory) et c'est un choix qui peut être **"override"** par un `config --local` (qui écrira l'information dans le répertoire `.git/config` du répertoire de ton projet). Donc oui, ce `git config`, tu fais ça une fois au tout début et ensuite, on n'en parle plus.
### Git - Les branches
- 1 branche = une ligne de développement = 1 suite de commits. La branche par défaut est `main`
- Une nouvelle branche est une **divergence** par rapport à la ligne principale de développement. Les branches coexistent dans la base de données. C'est à l'utilisateur de définir sur quelle branche il va travailler dans son espace de travail.
- L'étiquette `HEAD` est un pointeur qui nous permet de savoir sur quelle branche on se situe.
- Intérêt de plusieurs branches de développement :
- Au sein d'un projet collaboratif. Le développeur A peut travailler sur un fichier sur la branche A, et son collaborateur peut travailler sur un autre fichier sur la branche B, sans se piétiner (normalement, cf gestion des conflit à voir par la suite). Ils peuvent ensuite fusionner leurs branches pour mettre en commun leur travail, tout en conservant l'historique des modifications réalisées.
```bash=
git branch nombranch # permet de créer une nouvelle branche
git branch -d nombranch # permet de supprimer une branche (i.e., de supprimer son nom, l'étiquette, pas les commits)
git branch checkout nombranch # switcher de branche. Si la branche n'existe pas, elle est automatiquement créée
git switch nombranch # mise à jour de git = anciennement checkout
```
- Pour **merger une branche** : aller sur la branche cible où l'on souhaite intégrer les modifications.
- `checkout` == `switch` qui est présent dans les nouvelles versions de git.
- Dans les dernières versions de git `checkout` permet de revenir en arrière sur des commits à un instant t tout en conservant les versions ultérieure t+n.
> Ex: `git checkout id_commit`
- `git rebase` : peut être utilisé pour fusionner plusieurs commits en 1 seul commit
- `git fetch` : permet d'importer les modifications sans les fusionner à notre travail en local
> `git pull` = une fusion de fetch et merge
- `git log --oneline` permet d'affichier l'historique de façon plus compacte (voir aussi l'alias proposé plus haut et permettant d'avoir un `git hist`).
:::warning
**:warning: Bonnes pratiques :**
- Penser à effectuer des commits régulièrement
- Mettre régulièrement son travail en commun (merge)
- Standardiser les pratiques au sein de l'équipe
> Cela peut aussi
> passer par un langage commun dans les messages de commit. Suggestions en exemples: `git commit -m "del: message"` pour des suppressions, `git commit -m "up: message"` pour des mises à jour de fichiers existants, `fix:`, `clean:`, `add:`, `rename:`, `move:` etc. (il existe même des spécifications décrivant ce type de langage commun : https://www.conventionalcommits.org/)
- Récupérer ce qui a été fait avant sur le remote vers sa machine en local, `git pull`, avant de `git push` nos propres modifications sur le remote.
- Créer au moins un readme par projet (même un readme par répertoire)
:::



## Notebooks et literate programming
[GT Notebook](https://gt-notebook.gitpages.huma-num.fr/site_quarto/)
[Lien dépôt Literate programming](https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/literate_programming)
### Principe et historique
[Lien des slides](https://cours-376c29.gricad-pages.univ-grenoble-alpes.fr/#/title-slide)
- Sur la forge, possibilité de switcher entre source (.md) et visualisation en 'mise en page - rendered file'
- [Pandoc](https://pandoc.org/) : conversion d'une centaine de format en entrée vers une centaine de format en sortie.
> Exemple de customisation pandoc : [lien](https://learnbyexample.github.io/customizing-pandoc/)
>
<img src="https://dfzljdn9uc3pi.cloudfront.net/2017/cs-112/1/fig-3-full.png" width="50%">
- pandoc = **convertisseur universel** (md|docx|rst|org|... -> html|tex|md|org|...)
- Le format Markdown a été proposé et défini par John Gruber puis il y a eu plein de variantes autours de md (github markdown, ...). L'une de ces extensions est [CommonMark](https://commonmark.org/) et [Myst](https://jupyterbook.org/en/stable/content/myst.html) est un outil convertissant ce Markdown là vers plein de formats différents (html, doc, tex, typst, ...).
- [Tuto](https://e-publish.uliege.be/md/chapter/markdown/) rapide de prise en main du langage Markdown
> **Documentation** = comment un programme marche et comment l'interface utilisation devrait être utilisée.
> **Literate programming** = Ce qui nous a amené à faire ces choix en tant que développeur. :warning: Les seuls commentaires ne vont pas permettre de rendre compte de ce processus.
==Litterate programming== = Ne pas réduire le code à sa fonction computationnelle
Le format notebook permet de montrer les différents cheminements possibles pour aboutir à un choix de programmation. Peut être explicité au sein d'un `architecture.md`
https://www.reddit.com/r/programming/comments/le46br/why_you_need_architecturemd/
Pour plus tard : https://inacheve-dimprimer.net/
- `.ipynb` peut poser problème pour le versionnement car les résultats des calculs (les images en particulier, mais aussi les tableaux au format HTML) sont inclus dans le notebook:
1. Il est fréquent que ces sorties ne soient pas parfaitement reproductibles (quelques pixels qui changent de couleur, une taille de fonte un peu différente, ...)
2. Dans un format "binaire", il est extrêmement difficile de distinguer une différence *significative* d'une différence *non significative* (p.ex: un changement de taille de fonte), ce qui gène le suivi de version (de fausses nouvelles versions, plus de conflits difficiles à régler)
- Les notebooks sont souvent organisés de façon très linéaire alors que la programmation lettrée de Donald Knuth, qui met l'emphase sur le récit, permet des organisations non linéaires à l'aide de **chunks**. Étonnamment, même quand cette fonctionalité (de composition/narration non linéaire) est disponible (en particulier dans knitr), les développeurs ne l'utilisent pas, ce qui pourrait être interprété par le fait que les développeurs ne sont pas des gens qui aiment écrire des romans. :stuck_out_tongue_winking_eye:
> *NB : Dans la pratique, possibilité d'appeler les chunks au sein d'autres chunks*.
- Au passage, quand Arnaud dit que *The Art of Computer Programming* est illisible, on est sur une problématique un peu différente de la critique de [Programming Pearls](https://cours-376c29.gricad-pages.univ-grenoble-alpes.fr/#/polymorphisme-multiplicit%C3%A9-et-plurilinguisme-1) qui est très verbeux. La seule fois où j'ai plongé le nez dans ce livre, c'était pour en savoir plus sur des algorithmes de multiplication rapide de Coppersmith et Winograd et l'explication tenait en 3 exercices à faire par soi même et qui permettaient certainement de bien comprendre mais étaient inaccessibles au commun des mortels.
- Critiques autour du Notebook : ["The First Notebook War - Yihui Xie 谢益辉"](https://yihui.org/en/2018/09/notebook-war/#11-notebooks-make-it-hard-to-teach-well)
- Manuel [The turing way](https://book.the-turing-way.org/) pour une science des données reproductible, éthique et collaborative.
- [Software carpentries](https://software-carpentry.org/) = équivalent des ateliers de la donnée. [Lessons](https://software-carpentry.org/lessons/)
- Présentation de [litedown](https://github.com/yihui/litedown) : ["litedown: R Markdown Reimagined | Yihui Xie @ SOCEIO Seminar "](https://slides.yihui.org/2024-soceio-litedown.html)
### Mise en pratique - astuces
[Notebook & Programmation lettrée](https://mise-en-pratique-5e5223.gricad-pages.univ-grenoble-alpes.fr)
[Pratique individuelle](https://mise-en-pratique-5e5223.gricad-pages.univ-grenoble-alpes.fr/presentation.html)
[Lien vers formation DevSecOps](https://blog.stephane-robert.info/)
#### Job de déploiement
Le fichier `.gitlab-ci.yml` est créé pour être déployé à chaque modification du dépôt, i.e. à chaque commit.
:warning: Ce fichier `.gitlab-ci.yml` doit renseigner tous les dossiers/fichiers présents dans notre projet. Le job peut échouer si des fichiers présents dans le script n'existent pas dans le repot.
> Exemple : Si le fichier `biblio.bib` n'est pas présent il est nécessaire de supprimer la ligne `- mv biblio.bib public/` du script ci-dessous.
:arrow_right: Pour cela il est possible de modifier le fichier `.gitlab-ci.yml` proprement en local :
>
> `git pull` pour récupérer le fichier `.gitlab-ci.yml` créé sur le repo gitlab. Ouvrir le fichier `.gitlab-ci.yml` puis apporter les modifications. Puis coup de disquette (sauvegarde). Dans le temrinal : `git add .` en s'assurant que ce soit bien la seule modification ajoutée. Puis `git commit -m "up: message"`, puis `git push`.
```R=
image: rocker/geospatial:4.5
before_script:
- apt-get update && apt-get install -y curl wget git pandoc
- R -e "install.packages(c('quarto', 'sf', 'tidygeocoder', 'mapview', 'osrm', 'DT'))"
pages:
stage: deploy
script:
- rm -rf index_files/ index.html #dans le dépôt on a psushé ces fichiers mais on n'en a pas besoin donc on les dégage
- quarto render index.qmd #construire le fichier qmd, i.e. notre html
- mkdir public #créer un dossier que l'on va appeler public
- mv biblio.bib public/ # move ce fichier 'biblio.bib' dans public
- mv -v img public/ #on bouge le dossier image dans le dossier public
- mv -v *.html public/ # tout fichier se terminant par .html est bougé dans public
- mv -v index_files public/
- echo "Votre document a été déployé à ce lien $CI_PAGES_URL"
artifacts:
paths:
- public
only:
- main # nom de la branche
```
**Bonnes pratiques :**
- Commencer par créer un R.project
- Lors que l'on collabore sur la même branche : `git pull --rebase`. Recommandations sur l'utilisation de cette ligne de commande ?
[Pratique collective](https://mise-en-pratique-5e5223.gricad-pages.univ-grenoble-alpes.fr/presentation2.html)
## Reproductibilité computationnelle et contrôle des environnements logiciels
### Préambule et Introduction
Nous couvrirons une bonne partie du **[Module 2: Managing Software](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/objectives.html)** du [MOOC RR2: Practices and tools for managing computations and data](https://www.fun-mooc.fr/fr/cours/reproducible-research-ii-practices-and-tools-for-managing-comput/), sans faire tous les exercices bien sûr, mais vous pourrez du coup, facilement y revenir. L'enjeu est d'ouvrir la boite noire informatique et de reprendre le contrôle.
#### À faire, si possible, dans la nuit ou autre, avant le cours
Si vous avez installé `docker`, récupérez (`docker pull`) les deux images suivantes:
- [rocker/rstudio:latest](https://hub.docker.com/r/rocker/rstudio) (706.84 MB à télécharger, prendra 2.32GB sur votre disque)
- [rocker/geospatial:latest](https://hub.docker.com/r/rocker/geospatial) (1.55 GB à télécharger, prendra 4.92GB sur votre disque)
- [debian:stable](https://hub.docker.com/_/debian/tags) (ça, c'est tout petit)
- [ubuntu:questing](https://hub.docker.com/_/ubuntu/tags) (et ça aussi)
```
docker pull debian:stable
# Et pour ceux qui sont sous mac *et* qui ont un message d'erreur:
docker pull --platform linux/amd64 rocker/geospatial:latest
```
https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module0/seq3-sunspots/unit1-jupyterlab.html#windows
Petit problème avec WSL :
Vous tapez `wsl` dans le Powershell il ne se passe rien !
Peut être que la version s'est mal installée.
Faire un `wsl --list`
- si Ubuntu 24.04 n'est pas installé, installez le par `wsl --install Ubuntu-24.04`
- faire ensuite un `wsl --set-default Ubuntu-24.04`
Ensuite relancez `wsl` et cela devrait fonctionner...
#### Première petite expérience: Les Technophiles Anonymes
- Lancez R, chargez ggplot2 (`library(ggplot2)`), lancez `sessionInfo()` et indiquez ci-dessous la version de R et la version de ggplot 2 que vous utilisez:
```
R version 4.5.0 (2025-04-11 ucrt)
R version 4.4.2 (2024-10-31)
R version 4.4.1 (2024-06-14 ucrt)
R version 4.3.3 (2024-02-29)
R version 4.3.1
R version 4.5.1 (2025-06-13 ucrt)
R version 4.5.0 (2025-04-11 ucrt)
R version 4.5.0 (2025-04-11 ucrt)
R version 4.3.2 (2023-10-31 ucrt)
R version 4.5.1 (2025-06-13 ucrt)
R version 4.4.2 (2024-10-31 ucrt)
R version 4.3.1 (2023-06-16)
R version 4.5.0 (2025-04-11)
R version 4.5.0
R version 4.0.2 (2020-06-22) wow
R version 4.3.2 (2023-10-31 ucrt)
R version 4.3.1
R version 4.1.2 (2021-11-01)
R version 4.4.2
R version 4.5.0 (2025-04-11 ucrt)
R version 4.5.0 (2025-04-11)
R version 4.5.1 (2025-06-13)
--------
ggplot2_3.5.2
ggplot2_3.5.1
ggplot2_3.5.1
ggplot2_3.5.2
ggplot2_3.5.2
ggplot2_3.5.2
ggplot2_3.5.2
ggplot2_3.5.1
ggplot2_3.5.1
ggplot2_3.5.2
ggplot2_3.5.2
ggplot2_3.5.1
ggplot2_3.5.2
ggplot2_3.5.1
ggplot2_3.5.2
ggplot2_3.5.0
ggplot2_3.4.3
ggplot2_3.4.2
```
- Qui a déjà eu des problèmes pour installer du logiciel (R/python, bibliothèque R/python, etc.)?
> [name=Kim] Juste problème de droit admministrateur sur windows : nécessité d'exécuter les logiciels en tant qu'admin.
- Et pour cette école, malgré les efforts **hallucinants** des organisateurs ?
Ce n'est pas qu'une question de technologie, c'est les gens qui sont derrière : le soin apporté
#### Enjeux de la séance
- La base de l'installation de paquets sous linux.
- S'y retrouver un peu: Ubuntu vs. Debian vs. Pip/Condas/... vs. Renv/PipEnv vs. Docker/Singularity vs. Guix vs. ...
- renv/venv, setuptools/poetry/UV (wheel: binaire précompilé)
- Installer des logiciels en étant sûr de ne rien casser sur votre système, exécuter ces logiciels en isolation, et faire le ménage quand on en a besoin.
- S'assurer qu'on exécute bien tous la même chose
- Savoir construire soi-même un environnement logiciel de façon à peu près reproductible
- Prendre conscience de vos dépendances et développer un discours critique sur la modernité:

### Début de prise de note :point_down:
#### Reproductibilité computationnelle et contrôle des environnements logiciels
[**séquence 1**](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq1-sw_env_intro/unit1-lecture.html)
[slides](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq1-sw_env_intro/unit1-lecture/slides.pdf)
*Argh... damned computers*
" c'est pour cela que je ne mets pas à jour ma machine, parce que à chaque fois ça pète !" - reproduire le code de l'autre peut être difficile pour plusieurs raisons
- La *stack* logicielle et ses faiblesses

Au delà du fait de ne pas pouvoir reproduire un code d'une machine à l'autre, les maj des dépendances peuvent conduire à des changements dans les algorithmes des fonctions qu'on peut utiliser en shs. Notamment des fonctions où certains paramètres sont définis par défaut (ex: rnorm, centrée 0 variance 1)
L'environnement logiciel peut évoluer sans qu'on le contrôle.
- Variabilité d'un os à l'autre, par exemple (*horror stories*) :
*Free Surfer* (traitement d'image) avec différentes versions sur les différents OS : d'une version de FS à l'autre et d'un OS à l'autre on voit des grosses variations, ça pose problème quand on parle de tumeurs du cerveau à retirer (variation d'un facteur 1 à 2).
- Impact du compilateur (le compilateur, c'est ce qui transforme le code en langage binaire - une commande R a besoin d'un compilateur pour être exécutée
> Un **compilateur** sert à traduire un code lisible par l'Homme en code exécutable directement par la machine, dit "code binaire"
Par exemple, pour faire la différence entre un "wrapper", c'est à dire un code source et un binaire, il est possible d'écrire les commandes suivantes dans le terminal
- `cat /usr/bin/R` - code lisible par l'humain (source)
```{bash}
#!/bin/bash
# Shell wrapper for R executable.
R_HOME_DIR=/usr/lib/R
if test "${R_HOME_DIR}" = "/usr/lib/R"; then
case "linux-gnu" in
linux*)
run_arch=`uname -m`
case "$run_arch" in
x86_64|mips64|ppc64|powerpc64|sparc64|s390x)
libnn=lib64
libnn_fallback=lib
...
```
- `cat /usr/lib/R/bin/exec/R` - code directement exécutable par la machine (binaire)
```{bash}
@@@@��� ((�-�=�=hp�-�=�=�888 XXXDDS�td888 P�td DDQ�tdR�td�-�=�=HH/lib64/ld-linux-x86-64.so.2GNU��GNU�tPp��W
@ wAph7aOu�o`GN��0
�=�mj Cֺ�|CE��j�|2b��H�m��qX�D�
:�
�K�� bFV, |�"�@��(@� @�`"��"���� @����� @� _ITM_deregisterTMCloneTable__gmon_start___ITM_registerTMCloneTableRf_initialize_RRf_mainloopR_running_as_main_program__libc_start_main__cxa_finalizelibR.solibc.so.6__main__data_start__bss_start_end_edataMAIN_MAIN___IO_stdin_usedGLIBC_2.34GLIBC_2.2.5�����u�i �p�0
```
Exemple de variabilité des programmes compilés en fonction des options d'optimisaton du compilateur : le cas du calcul intensif (HPC) en astrophysique qui conduit à des erreurs d'un ordre de grandeur pour calculer la masse d'un objet/amas stellaire
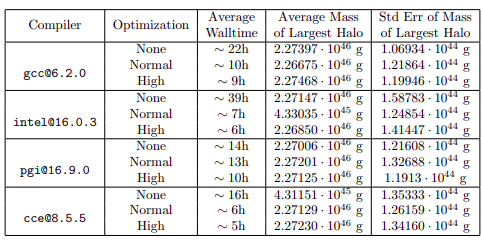
- Pour les langages interprétés (R, python..)
Variabilité dans l'environnement : un même *package/librairy* peut avoir plusieurs versions.
Des dépendances de python ou R sont écrites dans d'autres langages ([langage de "glue" - Glue code](https://en.wikipedia.org/wiki/Glue_code)). Ces codes sources ne sont pas visibles quand on cherche à retracer les versions des dépendances depuis le terminal R/Python
:::warning
:warning: **Bonne pratique**
- Renseigner les informations de la session à la fin du NoteBook avec les versions des packages utilisés avec par exemple `sessionInfo()`
- Prendre conscience et expliciter le processus d'installation des dépendances pour faire tourner le code
- Au delà des dépendances, expliciter la version du code que l'on fait tourner (`git log`)
:::
:::success
**Résumé** - Exécuter un code dépend :
- du code source
- des dépendances
- du compilateur/interpréteur
- des options de compilation
- des variables d'environnement
:::
- Anatomie d'un logiciel
Un code va rarement tourner seul. :arrow_right: `library(ggplot2)` : au début du code, on appelle des dépendances.
Pareil pour *bash*, les fonctions sont stockées en local, dans une certaine version. Il y a des variables d'environnement dont le(s) chemin(s) qui sont précisés dans la variable `PATH` en bash (écrire dans le terminal `echo $PATH`), qui permet de retrouver l'adresse du fichier binaire de ces fonctions.
> 🔎 Introduction sur les variables environnements : pour [ubuntu](https://doc.ubuntu-fr.org/variables_d_environnement), et pour [windows](https://www.malekal.com/variables-environnement-windows/)
*Fig - Exemple de résultat de la commande `env` dans le terminal*
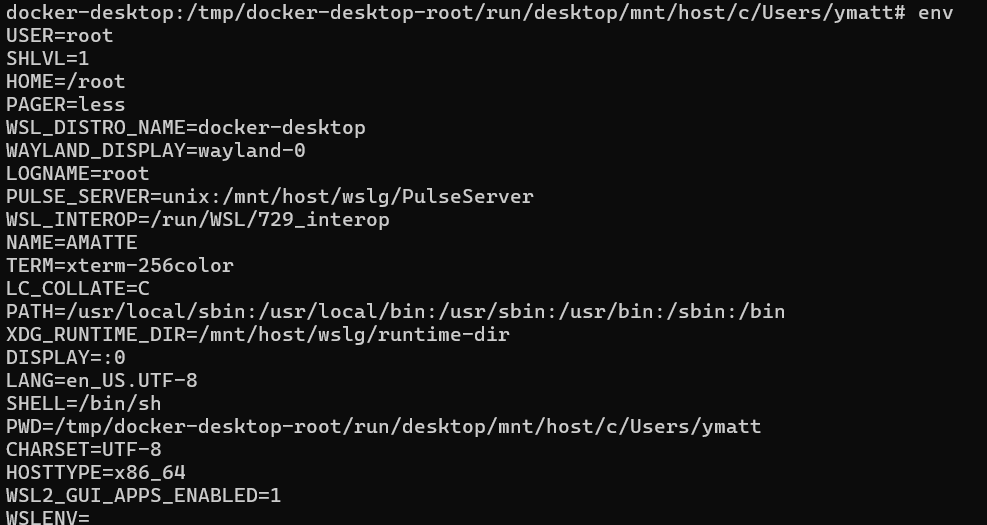
Les logiciels qu'on utilise vont avoir accès aux variables d'environnement pour pouvoir appeler des dépendances.
Si on utilise ces variables d'environnement, c'est pour avoir un environnement "unique", vers lequel vont pointer tous les logiciels. Facilite la maintenance
Pour savoir quel logiciel R on utilise,
- écrire dans le terminal `which R`,
et pour lire le programme qui *wrappe* ("emballe") le logiciel R,
- écrire `less /usr/bin/R`
puis pour connaître les dépendances de R (exécutable dynamique) - le binaire R,
- écrire dans le terminal `ldd /usr/lib/R/bin/exec/R`
Pour savoir quel logiciel *python* on utilise,
- écrire dans le terminal `which python3`
et connaître les dépendances de python (reporter le résultat de la commande précédente)
- écrire dans le terminal `ldd /usr/bin/python3`
[*Cheat sheet des commandes*](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq2-package_mgmt/unit2-references.html)
Dans tous les cas (langage compilé, interprété...), l'idée est de partager l'espace. Les librairies/dépendances peuvent être stockées n'importe où sur le disque.
**L'installation de logiciel fonctionne en trois étapes ==à partir de source==**
- Configure :
- Qu'est-ce qu'il y a comme bibliothèque sur cette machine ? Vu que ces libs sont dispo, je sais que c'est dispo.
- Make :
- Compile et lie les fichiers, crée une librairie partagée et exécutable
- Make install :
- Copie les libs partagées et exécutables
- droit administrateur aka `root`
- Permet aux utilisateurs autorisés d'exécuter le programme
:warning: ça peut créer des incohérences.
Je peux compiler un R, puis un deuxième. Les deux programmes vont être installés au même endroit :arrow_right: Conflits de nommage, peut modifier les dépendances, et casser les programmes préexistants.
**Installation des packages binaires (as root) sous ==Linux debian==**
:arrow_right: Installer non pas mais un programme déjà compilé, mais les fichiers sources. Permet de sauter la partie Configure / Make. Passe directement à Make install.
Problème : on saute le processus de suivi des libs déjà installées sur l'ordi.
`dpkg` : Permet de suivre les versions déjà installées sur l'ordi. Evite de réécrire des libs.
Si libs déjà installées, interdit l'écriture.
`apt` : A la liste de tous les paquets *Debian* et les télécharge tous. Quand on installe une librairie, permet de savoir quelles dépendances sont nécessaires. [lien](https://www.youtube.com/watch?v=ekr2nIex040)
Peut renvoyer un message d'erreur si on est pas autorisé à installer le paquet. Il est possible de contourner cela avec la ligne de commande `sudo <command>`.
[Tous les éditeurs de packages ne font pas ça : `pip` est une grosse brute, `conda` un peu moins]. Va réinstaller toutes les dépendances.
Les étapes 1 et 2 (`Configure` et `Make`) sont déléguées à quelqu'un, la maintenance du package. Système de confiance entre les utilisateur.rices et l'équipe de dev de la lib.
Où est-ce que `apt` sait où installer les paquet ?
`apt update` : quelles sont les dernières versions qui sont disponibles ? Stocke l'état actuel des paquet. à partir de là, si on installe, on installe la dernière version du paquet.
~ `git pull`
:::success
Pourquoi le focus sur Debian ?
- Open source leader (GNU/Linux)
- Communauté très grosse et ancienne, c'est carré !
- La reproductibilité sous Windows ou Mac Os est une boite noire.
- Depuis 1993 ça tourne toujours et très bien
- Question de sécurité : ça tourne partout et puisque c'est partout, c'est risqué. C'est un sujet qui a été pris au sérieux par la communauté Debian.
- Effort pour avoir des compilations reproductibles.
- Snapshot des archives des serveurs : on peut avoir accès au paquet Debian qui existait il y a 20 ans.
La formation historique GNU/Linux Debian d'Alexis de Lattre est encore disponible [ici](http://gabestock.free.fr/Sites_web/FormationDebianADelattre/) mais il faut l'adapter à la nouvelle version stable.
Pour une version (beaucoup (beaucoup)) plus complète et beaucoup plus récente, voir [Les cahiers du débutant](https://debian-beginners-handbook.arpinux.org/bookworm-fr/les_cahiers_du_debutant.html) *« Debian sans se prendre la tête »* d'[arpinux](https://arpinux.org/).
Pour aller plus loin, moins à jour, [The Debian Administrator's Handbook](https://debian-handbook.info/browse/stable/).
:::
Un des pb avec l'installation via le code source, c'est que c'est pénible. On laisse à l'équipe de dev la maintenance des dépendances, qui le fait plus ou moins régulièrement.
Si jamais on veut installer un logiciel qui n'est pas sur Debian ?
Par exemple Mapsf (bouh)
- Soit faire confiance à l'équipe de dev
- Soit passer par une autre procédure d'installation (`install.packages()`)
- Soit contribuer soi-même à Debian !
Et en utilisant des procédures d'installation ad hoc ?
```bash=
install.packages("ggplot2")
## Ne check pas la version des dépendances
```
```bash=
conda install pandas-0.13.2
## C'est pas si simple de retrouver des anciennces versions de packages (dépend de l'archivage des versions chez conda ?)
```
:warning: `pip`, `mamba`et `conda` s'ignorent tous : ils écrasent les variables d'environnement des uns et des autres et c'est le dernier installé qui prend le dessus.
**Solution** : Utiliser des environnements de développement. (ex. `pipenv`)
```bash=
pipenv python3
### Va créer un environnement de développement python3, qui n'est pas la version de python3 présente sur la machine.
```
Savoir quelles sont les dépendances permet de savoir quelles sont les versions utilisées.
Suivre les dépendances n'est pas standard.
La plupart des OS ont été conçus pour avoir une seule version d'un programme et ne gèrent pas plusieurs versions en même temps.
:::warning
- [name=Timothée]
Nuance : des procédures d'installation comme `install.packages()` fonctionnent pour tous les OS où R est installé. `apt` fonctionne seulement pour les distributions Debian.
Si on est sur Debian, privilégier `apt`, mais d'autres choix sont possibles.
Egalement, pour la rédaction de procédures d'installation dans une doc ou autre, `install.packages()` ou `pip install` permettent d'avoir une procédure commune pour les différents OS.
:::
:::warning
- [name=Arnaud]
A propos de la création d'environnements de développement : il faut garder en tête qu'il n'y a pas de garantie qu'on ne sorte pas de cet environnement ; il est facile d'en sortir. Ca peut être une bonne pratique, mais uniquement si on est rigoureux.
:arrow_right: Containerisation (`Docker`). Permet d'isoler l'environnement de développement du reste de la machine.
:::
[Feuille de pompe des bonnes pratiques](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq2-package_mgmt/unit2-references.html)
#### Comment fonctionne un système d'exploitation ?
[Slides](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq3-isolation_and_containers/unit1-lecture/slides.pdf)
Si on dépend d'un programme pour la reproductibilité, on dépend d'une boite magique.
Pour comprendre un programme il faut pouvoir accéder au code source :arrow_right: Open source.
*De quoi a besoin un programme ?*
Un programme a besoin du fichier binaire confié à une RAM.
Le programme ne s'exécute pas tout seul, il peut lire des données CSV par exemple. Ces entrées sont stockées sur le disque dur.
Pour fonctionner, un programme doit donc chercher des fichiers stockés partout sur la machine, et peut donc lire tous les espaces de stockages de l'ordinateur.
On devrait donc vouloir séparer les infos utiles au lancement du programme des autres.
*Qu'est-ce qu'un kernel ?*
- Comment on fait pour que les processus arrivent à s'exécuter en même temps sur le même processeur, être stockés sur le même disque dur ?
- C'est le noyau (kernel) qui permet à ces différents processus de s'exécuter, qui va fournir les abstractions permettant de sortir du binaire.
- `Open` `Write` `Read` `Close` ... ne sont pas des fonctions, ce sont des **appels système**. Demande au noyau d'interrompre ce qu'il fait pour exécuter une tâche.
- Comment on accède à un espace de stockage ?
- C'est le noyau qui est responsable de l'attribution et du suivi des espaces de stockage. Il va dire qu'à tel endroit il y a de la place et va l'allouer à un fichier.
:arrow_right: Quand on demande à un programme de lire un fichier ou d'écrire, en fait on lance un appel système pour demander au noyau d'exécuter l'action.
:warning: core = coeur, kernel = noyau
Une **distribution Linux** est un noyau Linux + tout un tas de programmes
#### Comment marche un conteneur ?
[Slides](https://learninglab.gitlabpages.inria.fr/mooc-rr/mooc-rr2-ressources/module2/seq3-isolation_and_containers/unit2-lecture/slides.pdf)
D'abord, *c'est quoi une machine virtuelle ?*
Une *machine virtuelle* est un programme qui va simuler un système d'exploitation.
Quand on exécute un programme dans une machine virtuelle, il y a des programmes qui s'exécutent dans la machine virtuelle et dans la machine (?)
Une machine virtuelle, c'est gros.
Emule un OS en entier. C'est compliqué à partager.
Une alternative plus légère c'est les **containers** : fonctionne avec *Chroot* (`chroot`), *Namespaces* (`namespaces`), et *Control groups* (`cgroups`)
Le premier problème, c'est qu'on a pas moyen de séparer les différentes parties du disque dur. Un programme a accès à tout le disque.
- `chroot` : permet de masquer et d'interdire l'accès à certaines parties du *root* aux programmes. Permet de modifier la racine du système de fichiers. Permet de choisir les parties du disque dur que l'on veut rendre accessible.
Même si on installe un programme dans le container, il sera isolé.
- `namespaces` : Permet de choisir quelles sont les instructions qui vont être lues par les programmes
- `cgroups` : limite, comptabilise et isole l'utilisation des ressources
Mais lancer les 3, c'est l'enfer :arrow_right: containers vont permettre de traduire ces instructions en abstractions intelligibles par des humains.
Une image Docker contient uniquement le code utile (pas de noyau ni de drivers).
Les processus s'exécutent normalement. Ils touchent au même disque dur. On peut partager une image sans ouvrir de connexion réseau.
D'autres containers que Docker ? `Apptainer` (ex-Singularity)
**Liste des fonctions docker :**
```bash=
docker help
## Visualise toutes les commandes de docker
docker images
## Liste toutes les images docker disponibles sur la machine
docker run debian:stable
## Lance l'image debian:stable
docker run -it debian:stable
## Lancer l'image debian:stable et permet d'interagir avec elle dans le terminal
## Pour sortir de l'image Docker, exit
docker build
## Permet de construire un docker file avec le suivi des changements effectués sur l'image
docker inspect
## Informations détaillées sur objets docker (conteneur ou image)
docker search
## Rechercher des images sur Docker Hub
docker run -e xxx
## crée une variable environnement
exit
## pour sortir d'une image docker
```
Le comportement par défaut d'une image Docker, c'est qu'elle est vierge quand on la lance.
Elle est isolée, donc n'a pas d'accès au disque de la machine.
Mais Docker garde une trace des modifications réalisées sur l'image.
non --> si tu fermes l'instance de l'image (le container) tu perds ce que tu as ajouté. En revanche il existe une commande pour sauvegarder ton container et créer une nouvelle image. Voir "docker commit".
[Software Carpentry Lessons](https://software-carpentry.org/lessons/)
Lancer RStudio (sur le port 8787):
```{shell}
docker run -e PASSWORD=toto -p 8787:8787 rocker/rstudio
```
Lancer RStudio, avec la stack geospatial et en mappant le répertoire de travail courant (de l'host) dans le dossier /home/rstudio/project (dans le conteneur), sur le port 8787:
```{shell}
docker run -e PASSWORD=toto -p 8787:8787 --volume=$(pwd):/home/rstudio/project rocker/geospatial
```
### Construire une image Docker
Créer /tmp/so-shs/ et /tmp/so-shs/docker. Copier le contenu ci-dessous dans /tmp/so-shs/docker/Dockerfile
```{Dockerfile}
FROM debian:stable
LABEL maintainer="Arnaud Legrand <arnaud.legrand@imag.fr>"
RUN apt update \
&& apt install -y python3
RUN apt install -y tint
```
Puis lancer (dans /tmp/so-shs/)
```{shell}
docker build -t alegrand/my_small_image:1.0 docker
docker images
```
alternative (dans /tmp/so-shs/docker/)
```{shell}
docker build -t alegrand/my_small_image:1.0 ./
docker images
```
```{shell}
docker run -it alegrand/my_small_image:1.0
/usr/games/tint
```
alternative (dans /tmp/so-shs/docker/)
```{shell}
docker build -t alegrand/my_small_image:1.0 -f ./Dockerfile .
```
L'image de Carl Boetiger: https://github.com/cboettig/noise-phenomena/?tab=readme-ov-file
Une alternative à https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/literate_programming/TP_published/mon_super_notebook_R
```{dockerfile}
FROM debian:stable
# FROM debian:stable-20250610@sha256:f06ef9fc5003fb8d069a67b4c4c752c9d47ba911ed77eba152de44e12798cac9
LABEL maintainer="Arnaud Legrand <arnaud.legrand@imag.fr>"
RUN sed -i /etc/apt/sources.list.d/debian.sources -e '/^URIs: /d' -e 's|^# http|URIs: http|'
RUN apt update -o Acquire::Check-Valid-Until=false \
&& apt install -y wget
# RUN cd /tmp/ && wget https://download1.rstudio.org/electron/jammy/amd64/rstudio-2025.05.1-513-amd64.deb && dpkg -i rstudio-2025.05.1-513-amd64.deb
RUN cd /tmp/ && wget https://github.com/quarto-dev/quarto-cli/releases/download/v1.7.32/quarto-1.7.32-linux-amd64.deb && dpkg -i quarto-1.7.32-linux-amd64.deb && rm -f quarto-1.7.32-linux-amd64.deb
RUN apt install -y r-base ### Boom! 250MB
RUN apt install -y r-cran-knitr ### Boom! 520MB
# RUN apt install -y r-cran-rmarkdown
RUN apt install -y r-cran-tidyverse ### Oh, only 3MB
RUN Rscript -e "install.packages(c('tidygeocoder','mapview','osrm'))"
RUN useradd --create-home --shell /bin/bash alvin
USER alvin
# RUN cd /tmp/ && wget https://quarto.org/docs/get-started/hello/rstudio/_hello.qmd && /opt/quarto/bin/quarto render _hello.qmd
```
Est-ce que cette image est reproductible ?
> Non car nécessité d'aller chercher un fichier à un endroit sans contrôle sur le contenu de ce que l'on télécharge.
> Manque de précision sur l'image debian

### Séance pratique / projet de groupe
Point de départ:
https://gricad-gitlab.univ-grenoble-alpes.fr/so-shs/literate_programming/TP_published/mon_super_notebook_R
À faire
- Compiler ce notebook sur votre machine (si vous y arrivez)
- Regarder le .gitlab-ci.yml et s'en inspirer pour compiler ce notebook sur votre machine, dans le même environnement.
- Y'a-t-il des différences entre les deux?
- Trouver le Dockerfile de Rocker, réaliser qu'on y comprend rien :stuck_out_tongue_winking_eye:, écrire un Dockerfile partant d'une petite image (debian:stable) et rajouter ce dont vous avez besoin (r-base, r-cran-knitr, le tidyverse, quarto, tidygeocoder, mapview, osrm).
- Prendre ainsi un peu conscience de ce qui se cache derrière tout ça.
- Rendre cette construction d'image aussi précise et reproductible que possible.
### Questions ouvertes dont vous aimeriez discuter
- Problèmes avec Docker-Desktop ?
- Docker in Docker vs. kaniko vs ... ?
## Vos remarques et suggestions
Nous aimerions recueillir vos remarques et suggestions sur cette école thématique. Qu'elles portent sur l'organisation, la forme ou le fond:
- Meilleure formation que j'ai suivi jusqu'à présent !
- Une semaine riche en connaissances. Presentations interessantes et utiles.
- Découvertes d'outils extrêmement utiles que je n'utilise pas, mais je compte m'y mettre après cette formation !
- Le contenu, extrêmement riche, pourrait facilement faire l'objet de plusieurs formations. J'ai appris énormement de choses !
Merci infini
Merci pour cette école. Semaine très dense où j'ai appris beaucoup de choses. Je pense pouvoir appliquer git dans mon travail.
J'ai bcp aimé les ateliers et la force de ces ateliers etait aussi le nombre d'encadrants qui permettait de ne jamais être bloqué.
- Retour sur les ateliers
- Bien que ça soit l'après midi.
- C'était le meilleur moment, l'heure passait vite.
- Fonctionnement avec les issues sur gitlab innovant et immersif.
- Penser plus les ateliers sur le contenu de la journée.
- Refaire une école ?
- Oui ! Mais pas forcément beaucoup de demande pour le moment ?
- Remarques
- Peut-être rajouter un atelier optionnel (comme celui d'Arnaud sur Emacs) de tutoriel autour des principales commandes PowerShell (malgré la diversité des OS) ou WSL/Ubuntu, car nous sommes un certain nombre à ne jamais l'ouvrir et donc à ne pas forcément le maîtriser. On a un peu appris sur le tas certaines commandes comme 'ls', "cd" ou "(mk)dir", mais il serait cool d'avoir un panorama des fonctions, car sans avoir d'informaticien ou une personne expérimentée dans nos groupes, c'était difficile de configurer le Shell.
Journées très complètes -> C'est bien qu'on finisse pas trop tard.
Le fait que vous soyez plusieurs animateurs permet à tout le monde d'avancer à son rythme.
Mélanger les niveaux, les instituts, c'est l'occasion de se rencontrer, de partager les pratiques et les points de vue, de s'entraider.
école très formatrice en pétanque, avec une forte montée en compétence des néophytes
- Axer l'école sur une technologie ? (Forge ou notebook ou docker)
- Non, c'était bien de présenter plusieurs outils. Dans mon labo, personne ne parle, ni de docker, ni de git et d'avoir une vue d'ensemble, c'était super. Ça permet d'avoir une bonne vue de l'écosystème de travail. Et même si on n'a pas tout dans les doigts à la fin, on nous a mis le pied à l'étrier.
- J'ai appris pas mal de concepts et dans telle ou telle situation, ça me sera certainement utile de savoir qu'il faudrait que je fasse comme ci ou comme ça, mais je ne suis pas encore opérationnel.
- A chacun selon ses besoins ? Faire des ateliers avec des niveaux de compétence différents afin que tous les profils s'y retrouvent, les débutants qui vont découvrir de nouvellles techniques comme les initiés qui sont là plutôt pour se consolider sur des points précis. Mais c'est sans doute dur à mettre en place.
- Finalement, même si on connaissait déjà les outils, de voir quelqu'un qui a du recul et qui présente bien, c'est des idées et des concepts dont on peut se saisir pour mieux expliquer les outils à nos collègues.
- J'ai été super satisfait sur ce format des ateliers. Il y avait énormément à faire, avec un socle minimal et des choses pour aller plus loin permettait à chacun de faire ce qui lui convenait. Rajouter plus de pratique pour s'assurer que chacun·e était opérationnel, n'aurait probablement pas été si pertinent que ça.
- Bravo et grand merci aux organisateurs et animateurs : c'est difficile de faire à la fois un panorama d'outils, tout en proposant des applications - le tout sans faire des journées à rallonge. C'était très réussi et très riche. Les ateliers à base d'issues sur gitlab étaient très bien conçus. Merci à ceux qui ont alimenté le pad. Seule suggestion : peut-être que faire des applications minimalistes en partant de zéro au lieu d'utiliser des fichiers déjà faits (pour notebook, intégration continue et dockerfile) serait une autre option. Plus charger les images docker et proposer des commandes pour tester la config wsl sous windows avant la formation. Encore merci!!
- Le pad collectif, pour que chacun partage la prise de note, c'est un très bon outil. On y ressent l'esprit collectif d'un labo et c'était inspirant. Continuer dans cette direction en prolongeant la prise de note après l'école ? La science ouverte, c'est un état d'esprit de collaboration.
- Pas assez de femmes.
- Est-ce que certain.e.s seraient intéressé.e.s pour faire réseau dans la suite de l'école ? Déjà commencer par une liste de diffusion ? à moindre effort, commencer par un chat ouvert à tout le monde pour partager des infos, histoire de voir déjà comment ça vit ? Plutôt que recréer un réseau, investir ceux qui existent déjà ?
- Le format des journées était très bien. Temps de pause réguliers, pour digérer (les infos). Site très agréable également, avec la possibilité de sortir de l'école. "C'était bien de finir à 18h, vous arrêtiez à chaque fois au moment où j'allais craquer." Avoir le temps de pouvoir se balader à vélo après et profiter du campus / de l'île.
- C'est ma 4ème école thématique et c'est la meilleure de 4 ! :smile_cat: Le fil rouge qui articule les thématiques permettait de revenir sur les points de bloquages.
- Les cours théoriques étaient vraiment bien et nous ont permis d'avoir des interactions allant au delà du champ disciplinaire.
- Avoir peut être des étiquettes nom car difficile de tout retenir ?
- Peur de ressortir de l'école "ébourrifé". Possible de faire un recap des technos et de l'articulation avec les sciences ouvertes ?
- Réseau Recherche Reproductible : c'est la meilleure façon qu'on a de tous monter en compétence. Pour creuser collectivement la question des sciences ouvertes en shs, il y a un intérêt à investir des réseaux.
- Merci à Franck et Arnaud, qui viennent pas des shs et permettent de transmettre des pratiques qui sont déjà intégrées dans leur disciplines.
- L'entrée logiciel aurait pu être plus explicite dans la communication de l'école:
- Ça aurait aussi pu couvrir les aspects données par exemple.
- Le fait que les gens montrent comment ils font, leurs propres pratiques et leurs limites, et pas une injonction institutionnelle, c'était très appréciable et adapté à des réseaux métiers.
- Il y avait beaucoup de formateurs, ce qui leur permettait de circuler et de prendre du temps pour aider chacun·e à aller à son rythme.
>"Je peux pas j'ai Rzine" - un tshirt floqué à faire ? :stuck_out_tongue_winking_eye: